美国NIH将建立人类生物分子图谱
变革性的技术让科学家能以前所未有的空间和分子分辨率构建组织的3D图谱。在未来7年,美国国立卫生研究院共同基金(National Institutes of Health Common Fund)人类生物分子图谱项目(Human Biomolecular Atlas Program, HuBMAP)打算通过支持技术开发、数据获取和详细的空间测绘,开发一个广泛可访问的框架,从而实现以单细胞分辨率全面绘制人体图谱。HuBMAP将与其他资助机构、项目、联盟和整个生物医学研究社区一起努力,实现一个全面、可访问的人体健康和各种疾病条件下的3D分子和细胞图谱的共同愿景。
人体是一个不可思议的机器。数以万亿计的细胞,跨越一系列空间尺度和多种功能状态,构成了人体的交响乐。虽然我们大致知道细胞在大多数组织中是如何组织的,但从组织和功能的角度对细胞和分子状态以及组织和器官中的相互作用网络的理解是非常片面的。不同类型细胞的3D组织结构以及细胞-细胞和细胞-基质相互作用,对不同组织和器官的正常功能、自然衰老、组织重塑和疾病进展具有深远的影响。近年来,新技术能够对多种细胞类型进行分子表征,允许研究人员以前所未有的规模和单细胞分辨率绘制它们在复杂组织中的空间关系。这些进展为建立高分辨率的人体组织和器官3D图谱铺平了道路。
HuBMAP (https://commonfund.nih.gov/hubmap)是一个由NIH资助的项目,其目标是开发一个开放性的框架和技术,在细胞分辨率下绘制人体的图谱,以及生成从不同年龄的正常人身上获得的几个组织的基本图谱。NIH之前资助的一个项目GTEx对大约1000个个体的DNA突变和大块组织的表达模式进行了检测,但HuBMAP是一个独特的项目,其重点是生成单细胞水平上分辨率的分子地图,但使用的样本来自更少的志愿者。为了实现这些目标,HuBMAP被设计成一个具有凝聚力和协作性的组织,具有开放和共享的文化氛围,使用基于团队科学的方法。HuBMAP组织(https://hubmapconsortium.org/)将积极与其他正在进行的计划,包括人类细胞图谱计划(Human Cell Atlas)、人类蛋白质图谱计划(Human Protein Atlas)、Lifetime项目(https://lifetime-fetflagship.eu/)、NIH相关组织支持的绘制特定器官(包括大脑、肺(https://www.lungmap.net/)、肾 (https://kpmp.org/about-kpmp/)和泌尿生殖系统(https://www.gudmap.org/)和组织(尤其是癌前和肿瘤组织(https://humantumoratlas.org)的分子图谱的项目,以及其他新兴项目进行合作。
HuBMAP的组织和方法
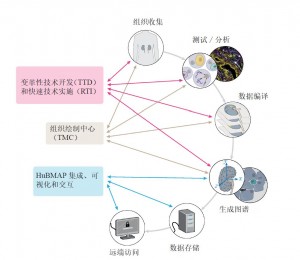
HuBMAP联盟由具有不同专业知识的成员组成(例如,分子、细胞、发育和计算生物学家、测量专家、临床医生、病理学家、解剖学家、生物医学和软件工程师、计算机和数据信息科学家),并被组织成三个部分:(1)组织图谱绘制中心(tissue mapping centres,TMCs);(2)HuBMAP集成、可视化和交互(HuBMAP integration, visualization and engagement,HIVE)协同;(3)创新技术组织(变革性技术开发(transformative technology development, TTD)和快速技术实现(rapid technology implementation, RTI))(图1)。在整个项目中,HuBMAP将会通过专项研究资金与其他NIH资金以及国际资金一起,协同性地扩大测绘的组织类型和技术研究。HuBMAP的后期阶段将会增加演示项目来展示生成资源的效用。更重要的是,HuBMAP将邀请更广泛的研究社区来分析HuBMAP数据以及来自其他项目或参与者自己实验室的数据。
图1. HuBMAP组织。TMC将收集组织样本,并生成空间解析的单细胞数据。参与TTD和RTI项目的小组将分别开发新兴技术和更成熟的技术。在今后几年,这些技术将大规模运用。来自所有团队的数据将由HuBMAP集成、可视化和交互(HIVE)团队提供给生物医学社区使用。随着图谱的逐步实现,这些小组将密切合作,以迭代的方式完善图谱集。
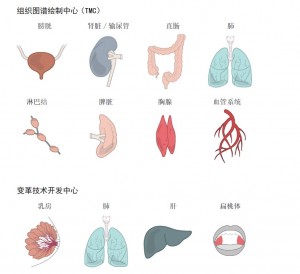
图2. HuBMAP最初分析的关键组织和器官。HuBMAP TMC将使用创新的、生产级技术为各种人体组织的单细胞3D图谱生成数据。与此同时,TTD项目(以及后来的RTI项目)将在一组差别很大的人体组织上改进测试和分析工具。该研究将包括来自不同性别和年龄的个体的样本。组织的范围将随着项目的进展不断扩大。
组织与数据生成
HuBMAP TMC将收集和分析大范围的正常组织,包括男性、女性、不同种族和不同年龄的成年人。这些组织(图2)包括(1)离散的、复杂的器官(肾脏、输尿管、膀胱、肺、乳房、小肠和结肠);(2)分散的器官系统(血管系统);(3)由具有不同微环境的动态或动态细胞类型组成的系统(淋巴器官:脾、胸腺和淋巴结)。收集组织时,HuBMAP TMC会精确记录解剖位置(如有可能,用照片记录),并根据已建立的方法,最大程度保护组织质量,减少降解。除了满足标准的监管要求外,还应尽可能获得同意,以便生成的数据可用于开放访问数据共享(即不需经数据委员会批准的公共访问),以便最大限度地为生物医学界所用。
为了实现空间分辨率的单细胞图谱,TMC将使用互补、迭代的两步方法(图3)。首先,进行组学测试,有效地收集数据,大规模并行地生成单个细胞的全基因组序列和基因表达谱。每个细胞的分子状态将由单细胞转录组和(在许多情况下)染色质可接近性测试来揭示;从开放的染色质数据结合基因表达数据可以推断转录因子结合区域,从而解释不同类型细胞中基因表达的调控。然后,通过成像方法(如共聚焦显微镜、多光子显微镜、光片成像等荧光成像方法)、序贯荧光原位杂交法(sequential fluorescence in situ hybridization, seqFISH)、质谱成像和质谱流式等技术获得组织或大块细胞中的各种生物分子(RNA、蛋白质、代谢物和脂质)的空间信息(丰度、特征和分布)。由此得到的广泛的单细胞和细胞核的信息将作为原位检测的提示(例如,单细胞或细胞核RNA测序结果将用于选择RNA或蛋白质探针),这将为多达数百个感兴趣的分子靶标提供空间信息。这些数据将允许对一个组织切片上的细胞进行表观基因组或转录组特征绘制,以揭示各种微环境状态。它们可能包括蛋白质在细胞质、细胞核或细胞表面的分布信息、磷酸化状态、复杂的组装、细胞外环境,以及蛋白标记共表达决定的细胞表型。复杂成像数据的配准和计算集成将提供超越任何单一成像模式的生物学信息。单细胞分析和多路原位成像的强大结合将为构建多种人体器官的多组学空间图谱及其在分子水平上的细胞互作打下坚实的基础。
TMC将使用互补的方法来收集数据,并强调通过流程来确保数据质量和元数据注释的标准化。在适当情况下,TMC将为方法学过程的每个阶段制定基准、质量保证和控制标准以及标准操作程序,并提供这些标准,以促进严格性、再现性和透明度。同时,生物样本和数据质量的保证和控制标准将随着组织收集、处理技术、存储和运输条件、分析技术和数据处理工具的改变以及HuBMAP与其他组织的合作,而不断进化。同时,HuBMAP会尽力标准化分析前变量的元数据(例如,注释和命名法)和技术,并与更广泛的研究社区共享方法和标准。
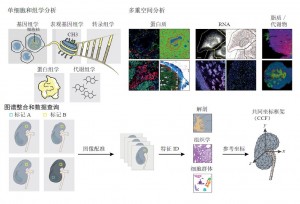
图3. 跨细胞和空间尺度的图谱生成和整合。HuBMAP的目标是生成一个图谱,用户基于其可了解来自器官特定部分的组织切片,并了解任意细胞多个“组”——基因组、表观基因组、转录组、蛋白质组和/或代谢组——的信息。为了达到这些目的,研究中心将结合成像、组学和质谱技术,以可重复的方式对来自人体特定部位的标本进行测试分析。然后,这些数据将被整合,形成针对任何特定组织的高分辨率、高信息量的3D图谱。为了确保个体间的差异不会与集合的异质性相混淆,HuBMAP将开发一个健壮的共同协作框架(common coordinate framework, CCF)。
构建完整组织图谱
HuBMAP产生的数据多样性,包括宏观和微观尺度(例如,解剖、组织学、细胞、分子和基因组)和多个个体,这些对其核心任务至关重要。通过对这些有价值的数据集的研究,我们将对人体有一个完整的认识。因此,HuBMAP将开发分析和可视化工具来连接空间和分子关联,以帮助生成高分辨率的人体3D分子图谱。
HuBMAP生成和收集的数据量需要使用、扩展和开发用于数据处理的工具和管道。虽然我们预计最初的数据处理工具将以联盟成员开发的方法为基础,HuBMAP也将与其他项目和更广泛的研究社区开发的算法一起工作和合并,以补充、增强或更新其技术管道。为此,HuBMAP将开发一个或多个门户,以适应通过一系列用户需求确定的新兴用例。这些开放源码门户将使用公认的标准,并可与其他平台(如HCA数据协调平台)互作,从而可以方便地添加、更新和使用新的软件模块(例如Dockstore和Toil)。HuBMAP的开放数据可以在多个平台上使用,也可以从多个平台访问,从而增强了其实用性。这个基础结构将使外部开发人员能够应用他们的代码、应用程序、开放的应用程序编程接口和数据模式,以促进HuBMAP数据与其他数据源的自定义处理和分析。此外,通过与其他全球和NIH项目的积极合作,HuBMAP将努力减少跨门户和平台浏览、搜索、聚合和分析数据的障碍。
通过将不同个体的空间和分子数据整合到一起,HuBMAP将创建一个通用的协调框架(common coordinate framework, CCF)。CCF定义了一个3D空间,利用早期的组织数据对技术和测试进行标准化。这种空间将作为所有HuBMAP数据的可寻址支架,支持统一的交互式探索和可视化(搜索、过滤和细节演示),并促进个人、技术和实验室之间的比较分析。为了实现这些目标,HuBMAP设想了一种策略,其灵感来自于其他组织图谱工作,利用“地标性”特征的识别,包括可以在所有个体中识别的关键解剖结构和组织的典型组成部分(例如,表皮边界和血管系统)。这些地标将使一个“半监督”的策略成为可能。该策略可用于校准和集成成参考数据,HuBMAP调查人员可以在此基础上施加不同的坐标系统,包括相对表示和基于区域的投影。例如,目前一个开源的、计算的组织学地形细胞分析工具箱(histoCAT)适用于二维可视化,不久也将适用于3D重建。同时,HuBMAP科学家也会探索基于本体论的框架,以便有效地对多尺度数据进行分类、导航和命名;预计这两种方法将产生协同效应。联盟将尽量收集和整合医学成像,如CT和MRI信息,将作为标记和构建CCF的基础。
技术开发与实施
对同一组织样本中不同种类的生物分子进行高空间分辨率、高灵敏度、高特异性和高通量的定量成像是构建详细的组织图谱的关键。虽然目前没有一种技术可以完全解决这一挑战,但互补功能的开发和随后的多路复用为加速组织定位工作提供了一种有希望的方法。HuBMAP创新技术小组的目标是开发几种创新方法,以解决现有最先进技术的局限性。例如,交换反应信号放大技术(signal amplification by exchange reaction, SABER)、seqFISH和Lumiphore 探针等变型技术将得到改进,以提高多个组织中RNA和蛋白质成像的多路复用、灵敏度和吞吐量。此外,新的质谱成像技术将能对来自同一组织切片的数百种脂质、代谢物和蛋白质进行高空间分辨率和敏感性的成像。该计划还包括开发和测试新技术。这些努力将受益于新计算工具和机器学习算法的开发。这些工具和算法首先基于项目早期一个常见组织生成的数据进行优化,然后用于跨尺度的数据集成。
挑战
以前的项目,如GTEx,面临的挑战是如何优化收集、保存和处理来自多个供体的各种组织类型。然而,HuBMAP的目标之一是使用各种各样的测试生成全面的、交互式的高分辨率图谱,这增加了复杂性。测绘一些具有重要功能的生物分子,包括一些我们可能甚至还没有意识到的分子,以及一些使用敏感性、特异性和高通量的测定方法仍然无法检测的分子,是一个需要特别关注的事情。此外,该计划将产生一个前所未有的数量和多样性的数据集,以用于全面的数据采集、管理、挖掘、建模、可视化探索和通信。需整合来自不同尺度(从细胞到组织,再到个体)的数据来生成稳健的图谱;必须开发相应的分析和交互式可视化工具,以确保整个生命科学研究社区都可以访问数据和图谱集。最后,考虑到人类图谱集的巨大规模,HuBMAP面临的挑战包括组织和技术的优先排序、跨组织和捐赠者的取样以及与国际努力的最佳协作。如果想要捕捉到罕见的细胞类型、状态以及组织结构,研究者需要研究多少细胞、视野和样本,这是一个难以确定的问题,但我们可以通过自适应功率分析法,利用来自HubMAP或其他联盟,或来自个人团体的、越来越多的数据来解决这个问题。
资源及社区参与
HuBMAP国际任务的一个重要组成部分是建立一个人体高分辨率细胞和空间图谱。我们坚定地致力于与各个研究团体展开协作,从而建立一个易于使用的平台和可互操作的数据集,最终加速实现高分辨率的人类图谱。围绕开放数据、工具和访问的共享指导原则将支持对不同财团产生的数据进行协作和集成分析。为了实现这种协同作用,HuBMAP和其他联盟将通过正式和非正式的聚会、基准测试、实践演示和研讨会,来共同应对常见的计算挑战,例如细胞注释。另一个潜在的紧密合作的实例是关于结肠研究:由HuBMAP、人类肿瘤图谱网络和Wellcome信托基金资助的多个项目将由Leona M.和Harry B. Helmsley慈善信托基金资助的项目来执行。由于项目侧重于部分不同的区域和疾病(例如,正常组织、结肠癌和克罗恩病),所有项目都必须确保以一致的方式收集和提供数据,HuBMAP将在这些工作中发挥积极作用。项目下一阶段,HuBMAP计划与NIH的其他项目合作,在2020年春季与人类细胞图谱计划(Human Cell Atlas initiative)举行一次联席会议,以确定和研究协调与合作的领域。与此同时,HubMAP的参与者也会参加其他协会,如人类细胞图谱或人类肿瘤图谱网络的会议和活动,从而形成紧密的联系。我们已经开始了一系列的开放会议来发展CCF,其中的第一次会议是最近与肾脏精准医疗计划合作举行的,会议重点是肾脏。
HuBMAP将提供数据提交、访问和分析功能,遵循公平(可查找、可访问、可互操作和可重用)的数据原则。我们将制定与类似举措相一致的、以常用格式及时发布和定期发布数据的政策。我们预计第一轮数据将于2020年夏季发布,之后会在固定的时间间隔内及时发布数据。稳健的元数据将包括标签和来源的所有方面,包括未识别的供体信息(人口统计学和临床)、组织处理和方案的细节、数据级别和处理管道。
的确,与更广泛的科学界和其他测绘中心进行接触和互动是确保HuBMAP产生的资源得到广泛利用并产生持续影响的关键。为了确保HuBMAP的浏览器和可视化工具是有价值的,该联盟将与解剖学家、病理学家、可视化和用户体验专家密切合作,包括那些在虚拟现实或增强现实方面有专长的专家。如上所述,我们希望本项目中包含的正常样本的多样性将有助于进行有价值的比较分析,精确地指出细胞和组织结构在个体、整个生命周期以及功能障碍和疾病出现过程中是如何变化的。该计划将根据这些实例构建其资源,并为与领域专家的密切合作提供未来的机会,例如演示项目。我们还预期这些数据将对产生新的生物医学假设、组织工程、时空交互的鲁棒模拟、组织特征的机器学习和教育目的非常有用。
结论
类似于第一个人类基因组图谱的发布,我们预期第一个3D组织参考图谱只是其最终范围和最终影响的冰山一角。HuBMAP通过与其他联盟密切合作,期望建立一个基金会,进而绘制正常人体的一个高分辨率的、关键器官图谱,并捕获个体之间的差异,同时该图谱还是不断增长的组织生物学和细胞生态学领域的重要资源。鉴于HuBMAP的重点是空间分子图谱,该联盟将为寻求类似目标的社区作出贡献,特别强调在开发其数据类型的分析方法和开发用于集成数据的公共坐标框架方面发挥领导作用。最后,我们希望催化组织结构的新观点,不仅是关于哪些类型的细胞彼此相邻,还包括定义这些细胞、其表型和功能相互作用的基因和蛋白表达模式。除了鼓励建立联盟内部和外部的合作,共同实现HuBMAP的总体使命,我们希望实现一个访问便利、公开可用的用户界面,可视化地展示单细胞层面的分子细节,感兴趣的分子通路和互作,以及一个给定的细胞类型随时间和空间发生的变化。研究人员还将能够浏览、搜索、下载和分析具有丰富元数据的标准格式的数据,后期用户还能够进行跨项目的数据集查询和分析。
重要的是,我们相信,该项目以空间解析的方式在单细胞水平上编译不同类型的多基因组信息,将是我们在理解人类生物学和精准医学方面迈出的重要一步。这些数据有可能重新定义细胞的类型或亚型,以及它们在组织内和组织间的关系,这超出了通过标准方法(例如,显微镜和流式细胞术)可以获得的传统理解。我们希望这项工作将为基于人体分子图谱的问诊、建模、导航和针对性的治疗干预奠定基础,这将是生物医学领域的变革。
原文检索:
HuBMAP Consortium. (2019) The human body at cellular resolution: the NIH Human Biomolecular Atlas Program. Nature, 574: 187-192.
张洁/编译