蛋白互动图谱有助阐明疾病原理
人类相互作用组:每一个点都是一个蛋白,每条线都是一个相互作用。
随着蛋白质相互作用研究的深入,研究者们现在可以解构精细的细胞内机制。
1987年,瑞士研究人员在论文中描述了两个具有相似异常的姐妹。这对姐妹小脑中的一片组织缺失,并且心脏上有小孔和裂缝。其中一个女孩在3岁时接受了心脏手术后死亡,另一个在4岁时也接受了类似的手术,但幸存了下来。因为这对女孩的父母都没有这些异常,研究人员得出结论,他们的女儿遗传了一个非典型基因的两个拷贝,导致患上以前未知的疾病。研究者们将这种疾病命名为Ritscher–Schinzel综合征。
这对姐妹所患疾病的突变可能存在于单个基因中。然而,研究者们后来发现,其它几种基因也与Ritscher-Schinzel综合征相关。这些基因的功能以及它们与Ritscher-Schinzel综合征的关联多年来一直是一个谜。
现在,蛋白质-蛋白质相互作用得到了系统性研究,并逐渐成为焦点,相互作用组领域随之兴起。通过蛋白质之间的连接网络的不断完善,三个研究团队各自发现了一种由突变基因产生的蛋白质组成的、名为Commander的蛋白复合体。Commander是细胞内的重要组成成分,负责对蛋白质进行分类和递送,Commander功能失调会导致Ritscher-Schinzel综合征。
蛋白质等生物分子很少“单打独斗”;大部分时候它们在短暂的互动中彼此作用,或者组合在一起形成复杂的分子机器。蛋白质只有通过这种合作关系才能发挥功能。这些相互作用的破坏可能会影响人体健康。
德克萨斯大学奥斯汀分校(University of Texas at Austin)的系统生物学家Edward Marcotte指出,如果编码一种蛋白质的基因发生突变,那么由这种蛋白组成的复合物在某种程度上也会功能失调,最终引起疾病或症状。
生物化学家早就开始研究一种或几种蛋白质与其它蛋白质相互作用的方式了。但现在,他们正在开发工具来系统地绘制从细胞器到生命体中的蛋白质-蛋白质相互作用的集合。通常,这些相互作用网络看起来就像星球爆发时的情景,蛋白质点或节点通过它们之间的相互作用彼此连接。这些网络中彼此作用的蛋白质可能代表关键复合体和共同功能,或,例如在Ritscher-Schinzel综合征中,为疾病根源提供线索。
在过去三年,研究人员发表了第一个高质量的人类相互作用组图谱。这些图谱确定了约93000个独特的蛋白质-蛋白质相互作用。
绘制这些图谱的技术并不新鲜——蛋白质相互作用的绘制可追溯到20世纪90年代。自21世纪初以来,研究人员一直在测绘蛋白组相互作用。但是方法学的改进以及蛋白质纯化、质谱和基因编辑技术的进步已经使研究人员能够以更高的精度探索相互作用组,并研究蛋白质互动在疾病发生发展中的作用。
然而捕获所有的相互作用是一件非常困难的事情,因为组成复合体的蛋白质可能分布在不同的组织、细胞,甚至存在时间也有差异。当细胞对环境进行响应时,蛋白相互作用是一个动态过程,时而连接在一起,时而分开。映射完整的蛋白相互作用组可能需要新的方法和系统生物学的思维方式。
尽管如此,该领域已经取得了一系列成果。Marcotte指出,虽然新的蛋白复合体层出不穷,但很多都被忽视了——这是互动组学图谱上延伸出的基础生物学。我们已经开启了一扇新的大门。
数字游戏
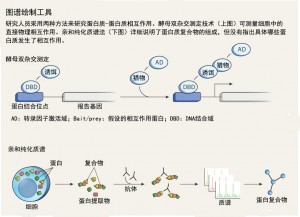
目前基本上有两种构建蛋白互动映射的方法。酵母双杂交测定法通过将基因表达与细胞中的蛋白质相互作用耦合,从而检测蛋白质组之间的直接互动。第二种方法则通过抗体分离出复合物,然后通过质谱法鉴定复合物的组成部分(参看文后“选择A或B”和图“图谱绘制工具”)来将测定蛋白之间直接和间接的互动。
Marcotte的实验室使用第二种方法的改进版,即使用蔗糖密度梯度等生物化学方法分离蛋白质,以了解哪些分子倾向于保持在一起。
Marcotte和他的实验室的博士后研究员Anna Mallam基于得到的图谱,对Commander蛋白复合体在细胞中的作用提出了假设。以前的研究表明,Commander的两个组分在结构上类似于构建和维持真核毛发结构的蛋白质;其它成分似乎具有跨膜运输蛋白质的作用。这些数据和其它研究结果表明,Commander将特定蛋白从细胞膜移动到高尔基体,在那里蛋白被回收利用。
最大的相互作用组学图谱包含成千上万的蛋白质,类似于星云。通过解读这些连接,研究人员发现了将癌症基因与“正常”基因区分开来的特征,并确定了关键的生物过程的特征,如细胞分裂过程中的染色体分离。
麻省波士顿Dana-Farber癌症研究所(Dana-Farber Cancer Institute)的计算生物学家Katja Luck也表示,即使采用多种方法,蛋白相互作用图谱仍然很不完整。这是一个数字的问题。人类基因组包含大约20000个蛋白质编码基因。如果假设每种蛋白质只有一种形式——事实上,很多蛋白存在多种形式——大约有2亿种可能的相互作用。实际数量可能会小得多,因为许多互动是间接的;一对一的互动估计有12万-100万个。
从生化的角度来讲,蛋白质是非常多样化的,因此它们的相互作用无法被每个测定法平均地捕获。例如,膜-蛋白质相互作用难以研究,因为当膜被剥离时,其形状和行为都会发生变化;它们可能会和平时的配体分离。但是,这种不完整性对当前图谱的影响尚不清楚。Luck指出,他们刚刚开始理解不同方法的偏倚。
作为遗传学家Marc Vidal实验室的博士后研究员,Luck已经改进了方法,消除了酵母双杂交方法中的错误。酵母双杂交法历史非常悠久,可以追溯到1989年。Luck指出,他们有做一些调整,以提高检测的准确性。通过用条形码标记蛋白质基因,团队可以在大范围的生长酵母中一次测试多个相互作用。严格关注细节、关键步骤自动化,以及4次测序,使他们能够识别6万个以上的相互作用,其中大部分是以前未知的。
该数据集构成了人类蛋白质相互作用组参考图谱(Human Reference Protein Interactome Mapping Project)的一大部分结果,而且这个数据集还在不断增长。Vidal表示,到2020年,他们可能需要一些可以作为参考的相互作用组学图谱。事情并不会总是一帆风顺。相互作用组领域在早期产生了一些错误的网络。根据2006年的一篇综述,只有约3%的、确定的相互作用得到了多种方法的支持。Vidal认为,尽管人们对使用这些数据集非常谨慎。但十年来,我们还是取得了非常惊人的进步。
更好的映射与CRISPR
Vidal的最终的参考图谱可能只是所有蛋白质相互作用组的一个子集。细胞和组织的变化以及细胞反应的变化,导致最后得到的互动组学图谱可能有多个版本。德国马普生物化学研究所(Max Planck Institute of Biochemistry)的生物化学家Matthias Mann认为,测绘这些变化是一件非常艰巨的事情。但他看好基因编辑技术(如CRISPR-Cas9)的潜力。
Mann的测绘方法包括建立表达数百种蛋白质的细胞系库,并使用名为Orbitrap的超高分辨质谱仪进行相互作用测试。诱饵蛋白与绿色荧光蛋白融合在一起,产生光度分布,从而得以让研究人员通过活细胞成像量化相互作用。Mann指出,在2009年左右,创建细胞数据库非常费力,CRISPR技术大大简化了这一流程。
自从2010年推出量化方法以来,Mann的团队已经绘制和量化了超过28000个互动。两个互动蛋白以一对一比例存在时,被认为是“强”互动,并且可能存在于稳定和丰富的复合物中。Mann表示,如果没有这样的信息,我们很难解析互动网络的结构。他的团队发现,人类互动体系由弱关联所主导,这可能提示,低丰度调节蛋白作用于更为稳定的蛋白。
细微的调整
整个领域的一个共同趋势是采用相对温和的样品制备方法,旨在真实地捕获细胞中的所有蛋白质-蛋白质相互作用。
加利福尼亚州生命科学公司Thermo Fisher Scientific的生物化学家Rosa Viner指出,他们正在努力寻找破坏性更低的方法。该公司专注于改进样品制备、工作流程和质谱技术,以帮助研究人员确定细胞中存在的相互作用。她补充:“这是最难的挑战——找到清晰度最高的方法,同时又不引入实验假象。”
这些实验假象包括在检测相互作用之前就离散开的蛋白质复合物。为了维持复合体的结构,Viner与加州大学尔湾分校(University of California)的研究人员合作,在质谱分析之前,对复合物进行化学融合——一种被称为交联的方法。这种策略名为QMIX(quantitation of multiplexed, isobaric-labelled crosslinked peptides,同位素标记交联肽定量),通过整合交联化合物与化学标记,稳定并跟踪蛋白质复合物。
良好的分析应该要考虑任何给定方法的盲点问题。波士顿哈佛医学院(Harvard Medical School)的细胞生物学家Wade Harper表示,有些类型的蛋白的相互作用研究起来非常困难。高通量分析的一个问题是,它会限制你对单个蛋白质的关注程度。这是因为这种分析倾向于对每个反应一视同仁,无法特别关注某些反应。
Harper和他的同事Steven Gygi也在哈佛带领一个团队来微调研究方法。Harper指出,他们团队比较小,只有四到六人。他们每个月可以创建四百到五百个细胞系。Harper等人由此创造了最大的、从单一流水线实现的人类蛋白质互动组学数据集。他们的图谱名为BioPlex,包括大约12万个互动。
前路漫漫
要深入了解蛋白互动,研究人员必须深入研究细胞。
加拿大多伦多大学(University of Toronto)生物化学家Anne-Claude Gingras使用了一种名为BioID的技术。该技术基于蛋白彼此之间的距离对蛋白进行标记。带有标记的目标蛋白质向附近的蛋白质添加了化学标签,以表示它与其它蛋白发生了相互作用。这就像握着蜡烛的小孩,走到哪里,哪里就会有光。
借助BioID,研究人员得到了围绕目标蛋白质的邻域图谱。据Gingras解释,鉴别蛋白质在细胞大环境(相比于几个蛋白之间的小环境)中的情况,有助于理解该蛋白的功能。
BioID的另一个优点是可以跟踪不能通过其它测定法检测的蛋白质,例如难以分离的膜包埋蛋白质。Gingras表示,他们和其他团队研究了染色质上的蛋白质,绘制了中心体组织,并检测了各种膜的相互作用。通过BioID技术,该小组在发育过程中发现了调节器官大小的信号通路中的新成分。
Harper实验室使用的是一种类似的方法——APEX。借助APEX,研究人员发现,名为抗坏血酸过氧化物酶(ascorbate peroxidase)的转基因植物酶调控着目标蛋白标记其它蛋白的时间窗口,从而得到更弱,但更空间上更精确的信号。
测定手段的多样化意味着由各种方法测绘得到的相互作用可以彼此印证。霍华德休斯医学研究所珍利亚研究园区(Howard Hughes Medical Institute Janelia Research Campus)的细胞生物学家Jennifer Lippincott-Schwartz指出,如果我们要了解细胞如何工作,那么将所有蛋白质之间的相互作用图谱与细胞内的空间图谱连接起来,至关重要。
细胞里有很多大结构或细胞器,它们全部漂浮在含有丰富蛋白质的细胞质中。要了解哪些蛋白质正在发生相互作用,以及为什么发生相互作用,这要求研究人员深入了解细胞环境。
Lippincott-Schwartz的实验室开发了一种工具,以用于使用荧光标记显现活细胞内的蛋白质。这些工具已经揭示了六个细胞器——内质网、高尔基体、溶酶体、过氧化物酶体和脂滴——的移动和相互作用3D模型。该团队称它为细胞器相互作用组。
Lippincott-Schwartz表示,互动组学是细胞生物学家的“假说生成器”。 如果你发现一个已知的蛋白和其它你不知道功能的蛋白在互动,那么你就该钻研下去,提出假设,然后去验证它。
随着蛋白质互动图谱越来越高精度,越来越完整,研究人员终于得以把假设付诸于实验。
更多阅读:选择A或B
两种高通量的蛋白质相互作用的测定方法各有支持者,但事实上这两种方法是互补的。
在物理层面,要确定两种蛋白质是否发生了相互作用,这需要借助酵母双杂交系统。该测定包括将编码两个被假设发生相互作用的蛋白的基因融合到酵母表达质粒的转录因子基因上,从而构成融合表达载体。只有当目标蛋白质相互作用,并结合活化关键基因时,这种菌株才能生长。对生长的酵母菌落进行测序,我们就可以反推出相互作用的两种蛋白质。
酵母双杂交系统允许一次快速筛选许多蛋白质对,但必须进行进一步检测来验证相互作用:两个蛋白质在酵母核中相互作用,并不意味着它们在其天然存在的细胞中也会相互作用。
研究人员还可以通过质谱来检测蛋白质复合物。质谱仪将复合体转化为带电粒子的云层,并通过其质量识别碎片。在一种常见的方法中,质谱分析前会进行亲和纯化——研究人员用蛋白质“饵料”标记肽或蛋白质。通过这些标记,我们可以从细胞浆中回收诱饵蛋白,或者随后进行质谱分析,以确定与这些“饵料”结合的蛋白。或者,研究人员可以从细胞中充分混合蛋白质,并通过一系列生物化学步骤进行分离,最后进行质谱分析。在这种方法中,一同被分离出来的蛋白质是相互作用的配偶体。
基于质谱法的方法允许研究人员直接在含有天然蛋白质的细胞中进行研究,而不需要在酵母中进行研究。但问题是,并不是所有的复合体都可以在提取步骤中维持稳定。此外,这些方法无法区分直接的物理交互、间接交互和松散的结构。
原文检索:
Marissa Fessenden. (2017) Protein maps chart the causes of disease. Nature, 549: 293-295.
张洁/编译