DNA错配影响蛋白质-DNA的结合
错配是DNA中的变化,它使双螺旋每条链上的碱基无法正确排列。由此可见,错配可以将DNA弯曲成有利于蛋白质结合的构象。
与DNA结合的蛋白质在生物学中普遍存在,这些蛋白质以高亲和力与特定的DNA序列结合的能力往往是其功能的核心,一个单一的突变影响蛋白质与DNA结合的能力并不罕见。令人惊讶的是,许多DNA结合蛋白可以更紧密地结合到那些被加工成含有一种名为“错配”(mismatch)的单核苷酸变化的序列上。这正是Afek等人在《自然》(Nature)杂志上的报道。
尽管突变和错配都涉及改变核苷酸的特性,但是二者之间有一个关键的区别。突变发生在DNA双螺旋的两条链上,这意味着维持了DNA每条链上的碱基配对。然而,错配只发生在一条链上,因此缺乏正常的碱基配对。在正常的碱基配对中,DNA双链中一条链上的腺嘌呤(adenine,A)碱基与互补链上的胸腺嘧啶(thymine,T)配对,鸟嘌呤(guanine,G)碱基与胞嘧啶(cytosine,C)配对,所以从A-T配对变为C-G配对是突变,而A-C则是错配。由于错配中碱基并没有配对,所以比突变更容易扭转DNA的整体结构(图1)。
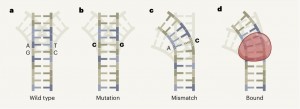
图1 DNA重塑。DNA双螺旋涉及成对的DNA碱基:一条链上的腺嘌呤(A)碱基与另一条链上的胸腺嘧啶(T)配对,鸟嘌呤(G)与胞嘧啶(C)配对。a,双螺旋可以存在一系列的形状,称为构象组合。在这个简单的示意图中,野生型DNA的主要构型在最前面,它可能暂时采用的次要构型是后面的米色阴影。b,突变将一对碱基变为另一对碱基(如A-T变为C-G)。这种突变不太可能改变可能的构象组合(尽管这种情况偶尔会发生,图中未显示)。c,在错配中,只有一个碱基被改变,破坏了碱基配对(例如,A-T可能变成A-C)。d,蛋白质的结合(红色)也可以改变DNA的构象。Afek等人报告称,10 %的错配将DNA弯曲成与野生型相比更为相似的与蛋白质结合的DNA构象,这使蛋白质更容易与DNA结合。
我们有理由认为DNA的扭转会损害蛋白质与其的结合,但事实上,它可以通过一种称为“形状读取”(shape readout)的机制促进结合的特异性。简单来说,形状读取是指蛋白质通过其特有的3D形状间接识别特定DNA序列的能力。这与它们通过直接识别每个碱基对中存在的特征化学基团的特定序列的能力相反,这种机制称为“碱基读取”(base readout)。DNA通常被认为具有相同的形状,无论其序列是否相同,但形状读取机制起作用是因为这不总是严格的正确。每个序列都有一组优先的构象,这种机制称之为“构象集合”(conformational ensemble),并且可以以不同的方式更容易或更不容易弯曲。利用这一点,需要与特定序列结合的蛋白质可以尝试弯曲任何它遇到的序列,使其与目的序列最相容。由于弯曲DNA有能量损耗,这种机制导致了蛋白质与DNA结合亲和力的下降。
形状读取机制在许多蛋白质和DNA相互作用中都起作用,但一直以来很难研究其真正的能量损耗,因为这样做需要干扰DNA分子的形状而不干扰其序列。Afek等人意识到,错配是DNA序列的微小变化,但却导致其形状有很大变化,这给来研究这种现象提供了一个独特的方式。
处理错配的DNA并不是一件小事,尤其是在高通量的情况下,因为许多标准的分子生物学技术都隐含着DNA是完全碱基配对的假设。因此,作者们开发了称为“饱和错配结合检测”(saturation mismatch-binding assay, SaMBA)的技术,它量化了在蛋白质与特定DNA序列中每一个可能错配的单核苷酸的结合数量。简而言之,他们制造了一种微芯片,其中排列有单链DNA,该单链DNA编码共有序列的每个可能的单核苷酸变体。每条链被放置在芯片上的一个已知坐标上。然后,他们使第二条互补的DNA链在阵列上流动。互补DNA与印在芯片上的每条链杂交,产生具有每种可能错配的双链DNA。最后,作者加入荧光标记的蛋白质,并使用显微镜观察其与DNA的结合。他们使用22套不同的DNA阵列和蛋白质进行了检测。
SaMBA发现,错配不仅有可能改善DNA与蛋白质的结合,而且错配也是比较常见的。在Afek等人分析的所有错配中,约有10 %的错配增加了蛋白质与该序列结合的亲和力,包括每个蛋白质至少有一个这样的序列。对于一些蛋白质来说,最有效的错配发生在天然的靶序列中,使蛋白质与该序列结合得更加紧密。对于另一些蛋白质,最有效的错配发生在非靶序列中,使蛋白质与该序列的结合水平与天然靶序列相当。在这两种情况下,同样的机制是占主导地位的:错配承担了扭转DNA的能量损耗,使蛋白质无需消耗能量。
请注意,要真正提高结合力,错配必须以与蛋白质通过形状读取机制相同的方式扭转DNA。以不同的方式扭转DNA会削弱结合力。错配也不应干扰蛋白质和DNA之间的任何化学接触,尽管作者确实发现,错配有时会带来有利的接触。
Afek等人的工作拓宽了我们对蛋白质如何与DNA结合的理解,并强调了DNA构象组合在这个过程中的重要性。将来,也许在自然界中不存在的核苷酸可以被用于SaMBA中,以充分探究DNA可以采用的构象阵列,这类似于非自然氨基酸被用于研究蛋白质生物物理学的细微变化的方式。SaMBA也有可能被改造为寻找旨在与不匹配或化学修饰的靶点结合的DNA结合蛋白,通过其它方法很难找到这种蛋白。鉴于大约三分之一的转录因子(一类调控基因表达的关键DNA结合蛋白)在人类中没有已知的靶序列,这可能是一个高产的研究方向。
更广泛地讲,错配通常能改善与蛋白质结合的发现可能对癌症等疾病有启示。即使是基因组中瞬时的错配,也可能促使转录因子结合在错误的地方,在那里它可能会错误地调控一个基因,使细胞处于癌变的转录状态,并且即使在错配被修复之后,这种状态也会持续存在。鉴于其“短期性”的根本原因,这种想法很难研究或证实。但错配改善蛋白质与DNA结合的明确倾向,使得这种机制值得深思。
原文检索:
Kale Kundert & James S. Fraser. (2020) DNA-binding proteins meet their mismatch. Nature, 587: 199-200. 郭庭玥/编译




