如何构建一个基因组
酿酒酵母(Saccharomyces cerevisiae)是第一批非细菌人工基因组合成项目的重点。
一套强大的分子工具可以帮助合成生物学家组装不同大小的DNA。
Leslie Mitchell本来并不打算做博士后,她原定的计划是,在渥太华大学(University of Ottawa)完成博士学位后,进入工业界。但后来,她的论文委员会成员、遗传学家Jef Boeke邀请她加入他在马里兰州巴尔的摩市约翰霍普金斯大学(Johns Hopkins University)的团队。Boeke正带头进行一项雄心勃勃的计划——从零开始设计和构建一个完整的酵母基因组,即Sc2.0项目。这是一个千载难逢的机会,她无法拒绝。Mitchell只是觉得这是学习生物学和真正理解它的最酷的方式,从头开始构建基因组。
8年后,Boeke实验室(现在位于纽约大学(New York University)的兰格尼医学中心(Langone Medical Center)),和他在欧洲、亚洲和澳大利亚的合作者即将合成全部16条酿酒酵母染色体的重新编码版本,以及第17条人工的“新染色体”。
迄今为止,只有少数基因组被合成,并且主要针对细菌。英国剑桥大学MRC分子生物学实验室(MRC Laboratory of Molecular Biology)的合成生物学家Jason Chin等人重写了大肠杆菌(Escherichia coli)的基因组,而美国克雷格·文特尔研究所(J. Craig Venter Institute, JCVI)的研究人员则构造了一个丝状支原体的“最小”基因组(Mycoplasma mycoides)(删除了所有非必需基因的基因组)。Sc2.0将合成真核生物(含有被核膜包围的核的细胞)的第一个基因组,这标志着DNA序列工程和组装的巨大进步。
英国曼彻斯特大学(University of Manchester)的合成生物学家Patrick Cai是Sc2.0的国际协调员,他表示,20年前,人们只是努力把几个基因组合在一起。现在,他们可以研究含有数千种成分的染色体。
用于合成基因组的工具和技术在更小的尺度上也被证明是强大的。例如,它们允许研究人员将定制的代谢途径串在一起,这样细胞就可以制造阿片类药物和抗生素。但细胞不像电路板那样容易重新布线,而且该领域仍无法实现其最终目标:设计出可预测结果的复杂生物系统。Cai指出,基因组设计的复杂性仍然比我们目前的工具所能支持的要高得多。
成本问题
在合成生物学的早期,研究人员面临着两个障碍:他们没有简单的方法来组装大量的DNA片段,也无法负担从商业制造商那里购买组件的费用。JCVI的合成生物学家John Glass表示,在世纪之交,一个定制DNA序列的收费是每核苷酸16美元。一个跨越几千个碱基的结构——大约是一个典型酵母基因的长度——价格超过5位数。
尽管现在染色体规模的DNA构建价格位于可接受范围内,但仍然不便宜。5年前,当Cai的团队第一次开始重建酿酒酵母的染色体时,他们向DNA合成公司支付了每个碱基30美分的费用。Cai指出,合成700个碱基大约需要20万美元的原材料。伦敦帝国理工学院(Imperial College London)的合成生物学家Tom Ellis则表示,现在类似的项目只需要不到一半的成本。
但目前尚不清楚,如果不重新发明合成技术,价格还能下跌多少。目前的主流技术是磷酰胺合成(phosphoramidite synthesis),已有几十年历史,它很难产生超过200个碱基的序列。任何更大的序列都必须通过将片段链接在一起来创建。
酶合成法是一种很有前途的替代方法。例如,在2018年,由合成生物学家Jay Keasling和他当时在加州大学伯克利分校(University of California, Berkeley)的博士生Daniel Arlow领导的研究人员演示了一种使用酶与核苷酸交叉连接的过程,尽管产生的序列只有10个碱基长。去年,总部位于巴黎的DNA Script公司宣布合成了一个200个核苷酸序列,这是迄今为止报道的最长的此类结构。其他几家公司也在朝着这个方向发展,包括2018年在伯克利由Arlow联合创办的Ansa生物技术公司,以及在加州圣地亚哥的Molecular Assemblies公司。Ellis认为,在5年内,我们将会看到与磷酰胺合成竞争的酶合成公司。
更大更好
研究人员现在经常将长达数千个碱基的DNA片段的生产外包给一些公司,如加利福尼亚旧金山的Twist Bioscience公司和爱荷华州的Integrated DNA Technologies公司。即便更大的DNA片段也能合成,但随着长度的增加,每一个碱基的成本会增加。Mitchell指出,这仅仅取决于你的银行账户里有多少钱,以及你想花多少时间来整理DNA。中国合成基因组学深圳重点实验室主任戴俊彪通常外包大约2000 – 3000个碱基的合成。据他估计,对于一个长达10 kb的片段,每个碱基的成本会增加一倍。戴会在自己的实验室里组装,因为他们经验丰富,而且他认为他们可以做得更快。
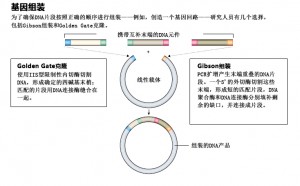
幸运的是,研究人员在寻找5,000到50,000个碱基之间的装配体时有几种选择。其中一个曾被用于组装最小的支原体基因组。Gibson组装是由JCVI的Daniel Gibson等人开发的,它利用的是两端有匹配重叠序列的DNA片段。一种外切酶被用来消化DNA的末端,并留下互补的单链序列。然后,其它酶以空缺处的另一条DNA单链为模板,将对应的脱氧核苷酸连接到单链上,并产生成品分子(图:基因组装)。
Gibson组装法可以在一个反应中有效地合成多达12个DNA片段,产生超过50kb的结构。但它在重复序列的组装上表现不佳,也不太适合构建多个小块DNA。英国诺里奇厄尔勒姆研究所(Earlham Institute)的分子和合成生物学家Nicola Patron表示,Gibson组装法在构建很长和很短的DNA序列上都不太行。Patron的团队大部分工作都围绕着结合多种基因和调控元素来改变植物细胞的功能。Patron发现,一种被称为“Golden Gate组装”(Golden Gate assembly)的方法可能更能满足自己的需求。
由合成生物学家Sylvestre Marillonnet和他在德国Icon Genetics的同事开发的Golden Gate组装体系,使用名为IIS型限制性内切酶的特殊蛋白质对DNA链进行有针对性的切割。这些酶被一个“识别序列”引导,但在距离识别位点一定距离的地方进行切割。研究人员可以定制由此产生的未配对末梢,这样不同的片段就可以按照规定的顺序组装起来。用户通常可以在一个反应中将5-10个片段组合在一起,形成跨越成千上万个碱基的片段。然而,对DNA切割酶的依赖意味着研究人员必须确保他们的片段都不包含不需要的识别位点。
合成生物学社区通过建立标准化部分的文库,包括基因、引导基因转录起始点的启动子序列和其他调控元件扩展了Golden Gate组装体系。Ellis指出,你可以像挑乐高一样挑选需要的组件。他的团队经常利用这个系统设计酵母基因回路,不同物种的Golden Gate试剂盒已经在实验室或商业化的地方共享,其中许多可以通过AddGene(萨诸塞州剑桥市的一个非营利试剂库)获得。Patron的团队开发了一个由大约350个部件组成的植物专用Golden Gate库,其他植物生物学团队非常看好这个库。Patron指出,她们的工具包已经分发给了200多个实验室,她猜每个实验室都至少制造了几个新部件。
创建染色体
Gibson组装和Golden Gate组装都具有成本上的优势——Ellis估计,一个典型的反应成本不到5美元。这些方法已经足够成熟,新用户不需要花很长时间就能上手。Patron表示,她们的实验室的一名本科生经过3周的培训,就能用Golden Gate组装体系组装多基因结构。
但对于长达数十万,甚至数百万个碱基的装配来说,挑战更加严峻。目前,唯一的解决办法是让活细胞来开展这一艰苦的工作。酿酒酵母具有高效的DNA重组机制,生物学家可以通过向细胞提供末端重叠的大片段来利用酵母的DNA重组机制,这和Gibson组装类似。这意味着研究人员可以利用酵母细胞将这些片段组装成长达100kb的长序列。哈佛大学(Harvard University)基因组学研究员George Church实验室的遗传学家Nili Ostrov指出,据他所知,所有大型合成染色体项目中都使用酵母体内组装。
装配通常是逐步实现的,这允许仔细的质量控制和故障排除。例如,戴的Sc2.0团队使用Golden Gate组装制造了中等大小的片段,然后按顺序重组成酵母染色体。他们将一次用3个10kb片段替换原基因组,也就是替换掉原基因组中的30kb长的序列。”
但是长序列很难处理。Glass提醒,当达到50、100或500kb时,难度会成倍增加。例如,常规的实验室程序,如移液器吸取操作,对只有几千碱基长的序列影响很小,但会对更大的片段产生破坏性的剪切力,破坏序列。
然而,逐步组装可以产生惊人的结果。最小支原体基因组包含超过100万个碱基。Sc2.0中正在构建的最长酵母染色体比这个大50%——大约1.5 Mb。中国科学院的研究人员成功地将酿酒酵母的整个基因组打包成一条长达近1200bp的染色体。
下一个前沿
到目前为止,大多数基因组合成的工作都集中在重写现有的材料上,而不是从头开始,但这些早期的尝试已经暗示了显著的基因组灵活性。例如,Ostrov等人已经开发出一种大肠杆菌的衍生物,这种衍生物的遗传密码仅使用了自然界中常见的64个3碱基构成的“密码子”中的57个,为重新利用剩余的7个密码子腾出了空间。她表示,她们倾向于把野生型作为基线,向左或向右移动一点,但也许她们可以尝试非常彻底的改变。
这反映了Cai在构建Sc2.0新染色体方面的经验。新染色体携带所有编码酵母菌转移RNA (tRNA)分子的基因,然后将这些基因从其他重新编码的染色体的原始位置删除。Cai解释,这些是基因组中的麻烦制造者,tRNA基因往往是基因组损伤和重排的位点。他们制造了新染色体,但新染色体仍然非常不稳定,因为他们“把所有的坏鸡蛋放在一个篮子里了”。
现在,该领域的先驱者正在超越成熟的酵母和大肠杆菌单细胞实验室模型,转向复杂的细菌——甚至进入植物和哺乳动物细胞。这些研究人员中的许多人已经在GP-write联盟的支持下联合起来。GP-write联盟是一个由Church、Boeke和其他机构牵头的国际机构,旨在简化与基因组设计和合成相关的成本和劳动力。戴正在中国协调一项类似的工作,它最初将专注于单细胞生物和病毒,但也在探索植物物种。
在去年10月发表的政策文件中,GP-write成员强调了该领域面临的技术挑战。最值得注意的是,这包括需要找到替代酵母的方法来组装大型DNA片段。这是因为在酵母中组装的染色体级别的DNA分子极其难以转移到其它生物体的细胞中,而且一些基因组不可避免地与酵母使用的机制不相容。目前还没有找到其它的寄主,但Glass怀疑还有其他寄主在等着被发现。Glass指出,很可能,大肠杆菌和酵母并不是自然界中最好的平台,它们只是目前我们所知道的最好的平台而已。
合成基因组的计算设计也是一个问题。马萨诸塞州波士顿大学(Boston University)的电气工程师Douglas Densmore在其职业生涯的大部分时间都在设计DNA回路,这种回路涉及相互连接的基因和调控元件,其原理与电子回路使用导线、电阻和电容器非常相似。Densmore指出,但与电子回路不同的是,DNA在蛋白质生成和结合位点等方面有更丰富的功能语言。
Densmore认为,由诸如Golden Gate这样的系统支持的模块化组装反映了电容和电阻是如何焊接到电路板上的。但是相对缺乏标准化的部件会使合成复杂化。设计工具是存在的,但它们会出现问题,因为这些库没有很好地定义、描述或模块化。由于基因调控的规则可能在一些细微,但重要的方面有所不同,因此这些平台在物种间的转化效果也不清楚。
Ellis表示,各个研究团队可以使用诸如基于云的软件基准测试等工具来计划和模拟Golden Gate组装和Gibson组装的装配反应。但这些工具难以应对大型项目的复杂性,因此迄今为止,染色体和基因组组装工作的大部分规划都还依赖于自制软件。为了让这类项目更加系统化和可预见性,Boeke在11月于纽约举行的GP-write会议上宣布发起一项为计算基因组设计开发软件的挑战,获胜者将获得“巨额现金奖励”。
提供的服务
Densmore认为自动化是一个宝贵的机会,可以使合成基因组的工作更加一致和可复制。他指出,让人们使用并遵守设计规则的最佳方法是让制造环节自动化。DNA组装背后的许多工作都是繁琐和重复的,许多小组发现把这些任务分配给机器人是值得的。
但建设内部自动化能力存在潜在的缺陷——尤其是从设备中获得的知识往往没有得到广泛分享。Patron表示,很多人都有这样的经历:你买了自动化设备,你那位了不起的博士后使用了它,你那位了不起的博士后离开了,然后没人知道如何使用这个仪器。因此,越来越多的机构已经建立了生物处理厂——学术机构可以用来进行DNA合成和装配的核心设施。其中一个例子是Earlham BIO合成中心,Patron在2016年帮助其成立。
目前全世界有20多家这样的机构,一些研究人员预计,该领域的大部分将依赖外包。Cai感觉,在未来几年内我们将能用电脑设计一段DNA并提交给合成中心,随后合成中心进行合成,第二天交付。Cai在2016年共同创立了爱丁堡大学基因组合成中心。
有了更多的工具,大规模基因组操作可能成为下一代研究人员的主流工具——可执行大胆想法的引擎,而不是目的本身。Cai的观点是,学者应该承担更多的风险,做一些创新性的事情,进入未知领域,而不是重复公司可以做,而且比学者做得更好的事情。
原文检索:
Michael Eisenstein. (2020) Building blocks for bespoke biology. Nature, 578: 633-635.
张洁/编译